paper: U-Net: Convolutional Networks for Biomedical Image Segmentation
code: PyTorch,官方公布的matlab的caffe版本
如果需要看网络结构,可以用这个prototxt文件unet prototxt,网络结构查看工具http://ethereon.github.io/netscope/quickstart.html
Abstract
- Unet是2015年的一篇工作了,在医疗图像分割中,能够在很少的训练集上实现比较好的分割效果。同时,设计的Encode-Decode结构(U形结构),在后来的很多方法中被借鉴使用
- 现在回过来看UNet的工作,整体思路还是比较简单的,具体的细节,将按照如下的网络结构图进行展开阐述:
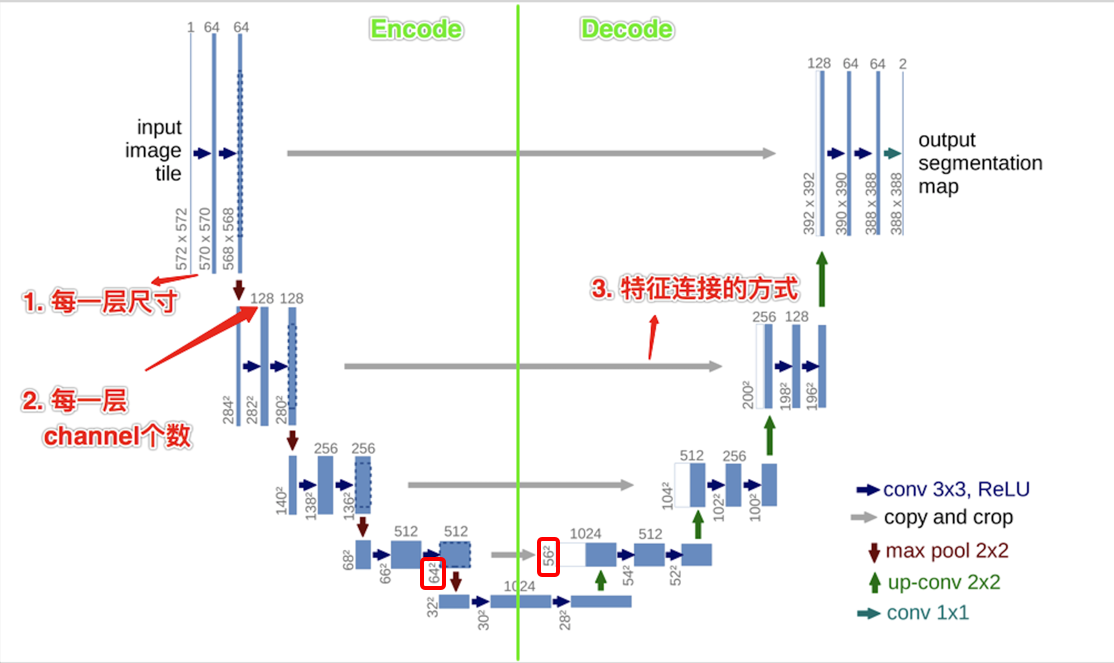
Details
- 每一层的尺寸
- 训练样本的大小是
512*512,为了能够包含到图像边缘的像素点,将训练样本扩充为572*572(图像边缘多30个像素,用图像内的像素对称填充),如下图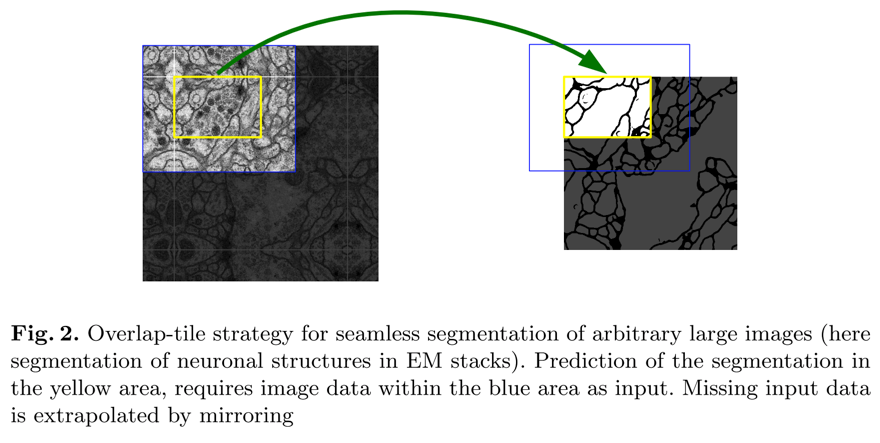
- 至于为什么扩充30个像素,个人理解是为了保证对Encode中的倒数第二个特征图(也就是Decode时进行concat的第一个层)crop时不会把原始图像的边缘信息裁剪掉,如上图中右侧原始图像的一个点,其按照Encode的反向过程计算的感受野大小为\( ((0\times2+2+2)\times2+2+2)\times2+2+2)\times2+2+2 = 60 \)
- Encode过程中,每一层的卷积都是valid卷积,即
padding=0,这就会导致每一层执行一次3*3卷积尺寸便缩小减小4个像素。这里如果用same conv,网络结构设计上应该也会更随意些 - 整个UNet除最后一层外全部使用的是
3*3(valid conv),最后一层使用1*1,共计23层conv
- 训练样本的大小是
- 每一层的channel个数
- Encode每次执行pooling后,kernel的个数都会增加一倍,而Decode的过程中,则是反过来的
- Encode-Decode结构的连接方式
- 这里连接是为了将低层特征和高层特征进行融合
- 对于低层特征(encode部分的feature map),由于前面提到的valild conv,在decode中的层执行upconv操作后,和原始尺寸还是有偏差的,因此直接将encode过程中的feature map裁剪成相同大小的feature map,然后在直接在channel维度concate,而不是element-wise的sum;同时,原始输入图像扩充,也保证了这里crop时原始图像的特征不会被丢弃
- Loss
- unet使用的是二分类交叉熵损失,如果用于多分类,该用多分类交叉熵即可
- unet本身对二分类交叉熵loss进行了修改,引入weighted map,使得网络能够学习到更好的边界信息,这里不再赘述。(个人理解,对于其他语义分割任务,用原始交叉熵即可)